Desde a descoberta de métodos para combater o ataque de agentes patogênicos (bactérias, vírus, etc..), ficou patente que os mesmos parecem desenvolver resistência contra medicamentos, pesticidas, etc. Será que esses organismos criam mecanismos inéditos para driblar os mesmos? Se sim, quais são seus mecanismos? Será que eles desenvolvem novos genes e traços fenotípicos para isso? Ou será que já posssuem mecanismos prontos para isso? Esse tema é assunto recorrente em discussões envolvendo a teoria da evolução, como podemos notar nessa publicação da autoria de Neil Greenspan, da revista The Scientist 1. Mas Palumbi já deixa claro o seguinte 2:
“In most cases, the causes of this evolutionary pattern are clear: if a species is variable for a trait, and that trait confers a difference in survival or production of offspring, and the trait difference is heritable by offspring […]”
Na maioria dos casos, as causas desse padrão evolutivo são claros: se uma espécie é variável para um traço (genético), e esse traço confere um diferencial na sobrevivência ou produção de descendência, e o traço é hereditário para a prole[…]
Ou seja, na maioria dos casos a causa da resistência se deve a traços e fenótipos já existentes na população de uma espécie, que podem se manifestar em certa parcela dos indivíduos, que, graças a isso, conseguem sobreviver e se reproduzir, ao passo que os que não manifestam esse traço acabam morrendo, e os sobreviventes que apresentam esse traço, se reproduzindo, dão origem a novas gerações cujos indivíduos irão manifestá-lo, o que se traduz em uma nova população resistente ao pesticida/antibiótico que antes era efetivo no combate à espécie.
Inclusive dois casos interessantes demonstram bem isso. Primeiro, foram encontrados corpos congelados de homens que participaram da expedição Franklin 3 4 (por volta de 1845), e em seus corpos, cepas de bactérias Clostridium, que foram ressuscitadas, e 3 dessas 6 cepas apresentaram a antibióticos criados mais de um século após a morte desses infelizes marujos, no caso, a clindamicina e cefoxitina! Outro caso 5 6 relata o seguinte:
WASHINGTON (Reuters) – Cholera bacteria and their cousins swapped genes to develop stronger, new strains long before antibiotics were invented, researchers said Thursday.
Bactérias da cólera e seus primos trocaram genes entre si para desenvolver novas cepas mais fortes muito antes dos antibióticos serem inventados, disseram pesquisadores, quinta-feira.
They said this showed antibiotics were not the primary driving force that has caused bacteria to mutate into ever stronger forms. Antibiotic-resistant bacteria are becoming a bigger and bigger problem. They range from penicillin-resistant gonorrhea to super-strains of staphylococcus that cannot be killed by vancomycin, the strongest antibiotic available. One of the things that allows bacteria to develop resistance to antibiotics is their habit of meeting and exchanging genes with one another.
Estes disseram que isso demonstra que antibióticos não foram a principal força que levou bactérias a mudarem em formas cada vez mais fortes. Bactérias resistentes estão se tornando um problema maior a cada dia. Estas vão de gonorreia resistente a penicilina a super-cepas de estafilococos que não podem ser mortas por vancomicina, o antibiótico mais poderoso disponível. Uma das coisas que permitem que bactérias desenvolvam resistência à antibióticos é seu hábito de se encontrarem e trocarem genes umas com as outras.
Didier Mazel and colleagues at the University of British Columbia studied the Vibrio genus of bacteria, which include the bug that causes cholera. They looked at samples of bacteria taken and preserved and dating back to 1888 — before antibiotics were discovered. They identified a mechanism of gene-swapping called VCRs, Vibrio cholera repeat sequence clusters, in the older bacteria. They look and act just like integrons, which bacteria are known to use to develop resistance to drugs. Integrons are used by cells to “read” new genes and convert them into working genes. “It is well established that integrons had a major role in the recent spread of multidrug resistance among Gram-negative bacteria,” they wrote in their report published in the journal Science.
Didier Mazel e colegas da Universidade da Columbia Britânica estudaram o gênero Vibrio da bactéria, que inclui a responsável por causa cólera. Eles observaram amostras da bactéria coletadas e preservadas desde o ano 1888– antes da descoberta de antibióticos. Eles identificaram um mecanismo de troca de genes chamados VCRs (sequências repetitivas da Vibrio cholera), nas bactérias antigas. Eles se parecem e atual igual a integrons, conhecidos por serem usados por bactérias para desenvolver resistência a drogas. Integrons são usados pelas células para ` ler” novos genes e convertê-los em genes funcionais. “Está bem estabelecido que integrons tiveram um papel importante na recente disseminação de resistência a múltiplas drogas entre as bactérias Gram-negativas,” escreveram em seu relatório publicado na Science.
Exatamente… Genes que causam a resistência à antibióticos modernos já existem nas bactérias há séculos/milênios (ou melhor ainda, desde o princípio…).. O texto acima apresenta um meio muito comum para a aquisição de resistência a antibióticos: a dinâmica troca de material genético entre as bactérias, mesmo entre as de diferentes espécies e entre bactérias vivas e mortas. Essa troca genética envolve mecanismos como a conjugação (a transferência de plasmídeos e transpósons por meio de contato direto entre as bactérias, ou através da transmissão de cópias genéticas por estruturas similares a agulhas), transdução (aonde a troca de materiais genéticos se dá por meio de vírus bacteriófagos) e transformação (aonde a bactéria pega DNA exógeno de meio externo, até mesmo de outras bactérias mortas).
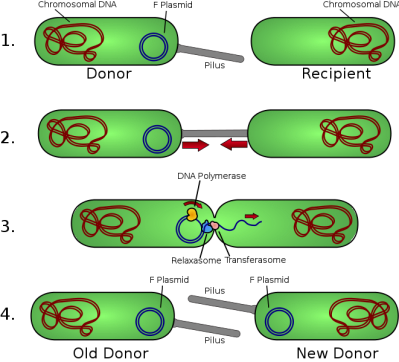
Essa troca de material genético é algo tremendamente corriqueiro e eficaz, ao ponto de ter sido relatado um caso intrigante de um paciente que fora infectado com uma cepa de Estafilococo:
” that was resistant to cadmium, penicillin, kanamycin, neomycin, streptomycin, tetracycline, and trimethoprim. Since each of these drugs operated by specific biochemical mechanisms that were used by a host of related drugs, the Australian staph could resist, to varying degrees, some thirty-one different drugs. In a series of test-tube studies the Australians showed that these various resistance capabilities were carried on different plasmids that could be separately passed from one bacterium to another. The most common mode of passage was conjugation: one bacterium simply stretched out its cytoplasm and passed plasmids to its partner.’” 7
“que era resistente a cádmio, penicilina, canamicina, neomicina, estreptomicina, tetraciclina e trimetoprim. Como cada um desses medicamentos operam por mecanismos bioquímicos específicos que são usados por uma série de drogas similares, o estafilococo australiano pôde resistir, em graus variados, a cerca de 31 drogas diferentes. Numa série de estudos os australianos mostraram que essa capacidade diversificada de resistência foi levada por diferentes plasmídeos que puderam ser transmitidos separadamente de uma bactéria para outra. O modo mais comum de passagem era conjugação: uma bactéria simplesmente estendia seu citoplasma e passava plasmídeos à suas parceiras.”
Outro mecanismo para a resistência bacteriana pode ocorrer por meio da modificação da droga usada, que passa a se tornar ineficaz no combate aos micróbios. A penicilina, por exemplo, pode ser inutilizada através da ação da enzima beta-lactamase, que quebra o anel β-lactama presente na penicilina e outros antibióticos. Fato descrito em um artigo da Science (link):
The most common mechanism of resistance in pathogenic bacteria to antibiotics of the aminoglycoside, beta-lactam (penicillins and cephalosporins), and chloramphenicol types involves the enzymic inactivation of the antibiotic by hydrolysis or by formation of inactive derivatives.
O mecanismo mais comum de resistência em bactérias patogênicas a antibióticos dos tipos aminoglicosídeo, beta-lactama (penicilinas e cefalosporinas) e cloranfenicol envolve a ativação enzimática dos mesmos através da hidrólise ou pela formação de derivados inativos deles.
Bactérias podem também receber de outras certos genes que codificam um produto bloqueado por certo antibiótico, como a sulfonamida, que bloqueia a síntese de ácido fólico, ao atacar certo gene. Recebendo genes diferentes que realizam a mesma função, através de plasmídeos, transpósons, etc, as bactérias afetadas podem tornar a sintetizar o ácido tranquilamente, tornando a atuação do antibiótico nula. Mas não podemos esquecer que a assimilação de plasmídeos advindas de outras bactérias impõem certos efeitos colaterais nas receptoras, diminuindo a taxa de crescimento e reprodução das mesmas. (Vide Bouma JE, Lenski RE (1988) Evolution of a bacteria/plasmid association. Nature 335: 351–352 [PubMed])
Bombas de efluxo
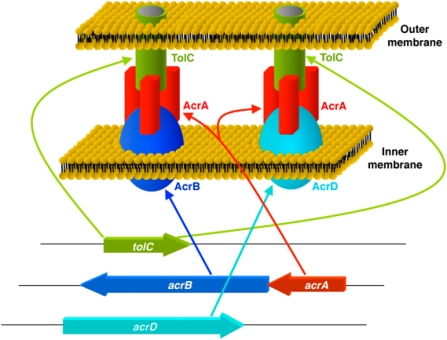
Existem também as bombas de efluxo, transportadores proteicos que ficam localizados na membrana citoplásmica, que funcionam através do consumo de ATP, e atuam na remoção de toxinas de dentro da célula (efluxo), ligando proteínas a molécula da respectiva droga antibiótica e levando-a para fora do interior celular por meio de exocitose. Esse mecanismo se assimila a atuação de outros componentes moleculares, tanto em procariontes quanto em eucarionte, como por exemplo, a P-glicoproteína em humanos, que consegue remover vários tipos de drogas anfipáticas das células.
Porinas e a resistência bacteriana
Bactérias gram-negativas possuem membranas externas que protegem as mesmas de agentes exteriores. Porém, certas proteínas chamadas de porinas agem transportando substratos de fora para dentro da célula (sendo certas porinas bem específicas, como a maltoporina, que transporta sacarose nas E.coli), e existem antibióticos que invadem as bactérias por meio dessas porinas. Mutações deletérias podem inutilizar genes que sintetizam essas proteínas, que falham em serem produzidas, e com isso, anulam a atuação de certas drogas que se ligam a essas, afim de invadirem a bactéria.
Certas drogas e outros substratos hidrofóbicos podem se difundirem diretamente pela membrana bacteriana, todavia sua entrada pode ser barrada quando ocorre alterações na bioquímica e composição das membranas, que não mais permitem a entrada delas na bactéria. 8
Mutações que levam à resistência bacteriana
É importante salientar que antibióticos atuam de inúmeras maneiras, seja atacando o DNA, causando danos diretos ao mesmo, ou prejudicando seu reparo (ao atacar e inutilizar as proteínas responsáveis por isso); seja prejudicando a produção de proteínas essenciais, perturbando sua forma específica (o que impede a mesma de se “encaixar” no seu alvo, como uma chave tem de se encaixar perfeitamente na fechadura para abrí-la) ou cessando sua produção; inibição da síntese de RNA; inibição ou perturbação da estrutura da parede celular, etc. Para isso, as moléculas da droga têm de entrar na bactéria e atingir o alvo específico que ela irá afetar. Elas podem fixar-se em proteínas transportadoras e, assim, adentrarem na bactéria, ou se fixarem em locais específicos no cromossomo, etc. E aí que entra a ação das mutações… Elas podem causar má-formação das proteínas transportadoras às quais as drogas se ligam, podem causar aumento vertiginoso na produção de proteínas atingidas pelas drogas, superando assim a ação destas, podem alterar a permeabilidade da parede celular, etc.
Um exemplo claro disso é a mutação no gene DHPS (responsável por produzir diidropteroato sintase. Neste caso, são duas mutações pontuais que ocorrem no gene.) que causa resistência á sulfonas como o sulfametoxazol (SMX) (fonte). O SMX funciona assim:
TMP and SMX, work sequentially to inhibit enzyme systems involved in the bacterial synthesis of tetrahydrofolic acid (THF)
TMP e SMX trabalham sequencialmente para inibir o sistema de enzimas envolvidas na síntese de ácido tetraidrofólico.
SMX is a structural analog of para-aminobenzoic acid (PABA) and competes with PABA to inhibit the synthesis of dihydrofolic acid, an intermediate step in the formation of THF [1,3]. SMX binds to dihydropteroate synthetase which catalyses this reaction
“SMX tem estrutura análoga ao do PABA e compete contra o mesmo para inibir a síntese de ácido diidrofólico.
Reduced availability of THF inhibits thymidine synthesis and subsequently DNA synthesis
Disponibilidade reduzida de THF inibe a síntese de timidina e subsequentemente a síntese de DNA. (Fonte)
Relatando a resistência contra penicilina em bactérias do gênero Streptococcus, o seguinte artigo diz:
β-lactam resistance in S. pneumoniae is a consequence of generating mosaic pbp genes. Such genes encode penicillin-binding proteins (PBPs) harboring tens of substitutions throughout the entire protein [22], which causes these genes to lose their affinity for the antibiotics.
Resistência a Beta-lactama em S. pneumoniae é consequência da geração de genes mosaicos pbp. Tais genes codificam PBPs (proteínas às quais a penicilina se liga) contendo dezenas de substituições (de aminoácidos) por toda a proteína, o que leva esses genes a perderem sua afinidade com os antibióticos. (afinidade refere-se à capacidade da droga em se ligar à proteína)
E:
“Generally speaking, mutations of these residues in the binding site could affect antibiotic affinity.”
“Falando de maneira geral, mutações desses resíduos (aminoácidos) na área de ligação poderia afetar a afinidade antibiótica” (Fonte)
Outros exemplos de como mutações barram a ação eficaz de drogas:
INH: Ou isoniazida, é uma substância que, uma vez dentro da bactéria, é ativada pela catalase peroxidase (codificada pelo gene katG), tornando-se uma toxina que afeta alvos como a síntese de ácido micólico, fundamental componente da parede celular. Carência desse ácido resultará na perda de integridade celular, ocasionando a morte da bactéria. Mutações no gene katG que causam a perda da atividade da catalase levam à resistência contra INH. Um dos alvos do INH é a proteína codificada pelo locus inhA, que participa da síntese do ácido micólico. INH se liga ao complexo InhA-NADH, formando um complexo triplíce que inibe a síntese do ácido. 6 mutações pontuais no gene inhA foram identificados como responsáveis por resistência ao INH;
RIF: Ou rifampicina, essa droga interfere com a transcrição do RNA ao se ligar a uma subunidade da RNA-polimerase, barrando a transcrição, o que culmina na morte da bactéria. 69 Mutações no gene rpoB (que codifica a Beta-subunidade à qual o RIF se liga) foram encontradas em M. tuberculosis resistentes a RIF;
PZA: Ou pirazinamida, tem como alvo uma enzima essencial para a síntese de ácido graxo. Sua forma ativa, POA, ocorre graças a PZase, codificada pelo pncA. Só funciona nas Mycoplasmas da espécie tuberculosis. A Mycobacterium bovis, por exemplo, é resistente ao PZA por possuir uma mutação no gene pncA (uma troca de Citosina por Timina). Na variedade tuberculosis, a droga só funciona em Ph externo ácido, que prejudica o funcionamento da bomba efluxo, permitindo que o POA se acumule dentro do citoplasma, reduzindo o Ph interno ao ponto de inativar uma sintase vital para a síntese dos ácidos graxos. Várias mutações no gene pncA que conferem resistência foram descobertas;
ETH: Ou etionamida, é uma droga que ataca a síntese de ácido micólico. Mutações no promotor do gene inhA são associados com a resistência ao ETH. E mais, o EthA cataliza a ativação do ETH em duas etapas; mutações que levam a redução na atividade do EthA, conferindo resistência. A expressão do EthA está sob o controle do gene vizinho ethR, que codifica um supressor que regula negativamente (isto é, reduz) a expressão do ethA.
Mais exemplos podem ser encontrados neste link.
Mutações aparentemente benéficas podem causar problemas e efeitos colaterais diversos nas mutantes:
Recent studies have demonstrated that antibiotic resistance, whether caused by target alteration or by other mechanisms, generally confers a reduction in fitness expressed as reduced growth, virulence or transmission. 8
Estudos recentes demonstraram que a resistência à antibióticos, seja ela causada por alterações no alvo atacado pelo medicamento ou por outros mecanismos, geralmente conferem uma redução no “fitness” bacteriano (aptidão, capacidade da bactéria em sobreviver, alimentar-se, reproduzir, etc), manifestada pela redução no ritmo de crescimento, capacidade de virulência (capacidade patogênica de um microrganismo, medida pela mortalidade que ele produz e/ou por seu poder de invadir tecidos do hospedeiro.) e/ou de transmissão (entre hospedeiros). 9
Poderíamos citar dezenas de estudos, mas, afim de não prolongar demais o já extenso texto, vou citar brevemente um artigo de 2007, publicado na Genetics, site pertencente à Genetics Society of America. 10 Nesse estudo os pesquisadores analisaram os custos no “fitness” de E.coli que adquiriram o gene blaTEM-1 β-lactamase, responsável pela resistência à antibióticos da classe β-lactâmicos, tipo a carbapenema. Alguns efeitos colaterais da aquisição do plasmídeo contendo esse gene são:
“Carbapenem resistance is often associated with decreased cell-wall permeability”
“Resistência à carbapenema é amiúde associada com reduzida permeabilidade da parede celular” (redução na capacidade da parede de absorver substâncias, incluindo nutrientes)
“Regardless of the cause, the association with a fitness cost may explain why AmpC-producing Salmonella are seldom isolated (MOROSINI et al. 2000). Subsequent work by HOSSAIN et al. (2004) found that another class C β-lactamase, CMY-7, impaired Salmonella typhimurium strain LT2 growth and cell invasion when encoded on a high-copy number plasmid.”
“Independente da causa, a associação com custos ao fitness podem explicar porque Salmonella produtoras de AmpC são raramente isoladas. Pesquisas subsequentes de Hossain e colegas (2004) revelaram que outra β-lactamase classe C, a CMY-7, prejudicou o crescimento e habilidade de invadir células da cepa LT2 da Salmonella typhimurium quando codificadas (elas, as lactames) em plasmídeos “high-copy number” (isto é, plasmídeos com vastas quantidade de cópias)”
“The observance of plasmid instability and cell lysis in overnight cultures containing plasmids encoding blaSME-1 suggested the possibility of a fitness cost associated with the SME-1 gene in E. coli.”
“O fato de instabilidades no plasmídeo e lise celular (A Lise Celular é o processo de ruptura ou dissolução da membrana plasmática ou da parede bacteriana causada por osmose, ataques virais, ação enzimática, etc, que leva à morte da célula e à liberação de seu conteúdo.) serem observadas em culturas de E.coli contendo plasmídeos codificando blaSME-1 sugere a possibilidade de custo ao fitness da bactéria associado com o gene SME-1 na mesma.”
“E. coli K12 XL1-Blue transformed with plasmid-expressing SME-1 β-lactamase (pTP123-SME-1) exhibited an extended lag phase and an overall reduction of the final cell density in stationary phase”
“E.coli expressando SME-1 β-lactamase exibiram prolongadas fases de “atraso” (fase durante a qual as bactérias acostumam-se com o meio onde se encontram e começam a multiplicar-se) e uma geral redução na densidade celular final durante a fase estacionária.”
Mais adiante os autores relatam um teste aonde colocaram E.coli com plasmídeo pTP123-SME-1 para competir com E.coli desprovidas do mesmo plasmídeo, em um meio de cultura contendo arabinose, por uma noite inteira. Passado esse período eles analisaram o meio e constataram que 98% (margem de erro de 3%) das bactérias presentes eram do grupo sem o SME-1!
Resumo
Diante destes e outros dados similares, conclui-se que a resistência contra antibióticos ocorre através de traços genéticos já existentes que são transmitidos entre as bactérias por inúmeros meios, através da ação de bombas de efluxo, alterações na composição da membrana celular e, finalmente, através de mutações que, como vimos acima, podem barrar a entrada de antibióticos, desativar enzimas às quais eles se ligam, mudar a permeabilidade da membrana celular, etc. Vimos também como nenhum gene ou função inédita surge, mas apenas a transmissão de genes já existentes entre as bactérias. Por fim, notamos como as ditas mutações benéficas trazem consigo a perda de “fitness”, exemplificado nos casos de crescimento prejudicado, redução na reprodução, problemas na permeabilidade da membrana e até mesmo lise celular (aonde a bactéria literalmente explode, devido à instabilidade de sua parede celular ou desbalanços osmóticos letais).
Referências
1 Greenspan, N.S., Not-so-intelligent design, The Scientist 16(5):12, 2002
2 Palumbi, S.R., Evolution—humans as the world’s greatest evolutionary force, Science 293:1786–1790, 2001; p. 1787.
3 McGuire, R., Eerie: human arctic fossils yield resistant bacteria, Medical Tribune, 29 December, 1988, pp. 1, 23
5 Rense http://www.rense.com/health/studysuperbgs.htm
6 HighBeam Business http://business.highbeam.com/435553/article-1G1-56448565/bacteria-swap-genes-become-more-resilient
7 Garrett, L., The Coming Plague: Newly Emerging Diseases in a World Out of Balance, Farrar, Straus, and Giroux, New York, 1994.
8 Chevalier, L, Pages, J.M., Eyraud, A. and Mallea, M., Membrane permeability modifications are involved in antibiotic resistance in Klebsiella pneumoniae, Biochemical and Biophysical Research Communications 274(2):496–499, 2000.
9 Andersson DI (2006) The biological cost of mutational antibiotic resistance: any practical conclusions? Curr Opin Microbiol 9: 461–465 [PubMed]
10 David C. M, O Y. Karkouti and T. Palzkill A Fitness Cost Associated With the Antibiotic Resistance Enzyme SME-1 β-Lactamase June 11, 2007 doi:10.1534/genetics.106.069443 (link)